解析の準備¶
ここでは、観測した生データのダウンロードと解析を実行する前に必要な準備について記載しています。
HSC 生データのダウンロード方法¶
Warning
ダウンロードの前に バイアス、ダーク、フラットデータについて をお読みください。
観測データは STARS (Subaru Telescope Archive System) からダウンロードできます。
STARS にログインすると、図1 のようなページに飛びますので、Search by タブから Proposal_ID Instrument や Proposal_ID を選択し、 自分のプロポーザル ID を選択してください。
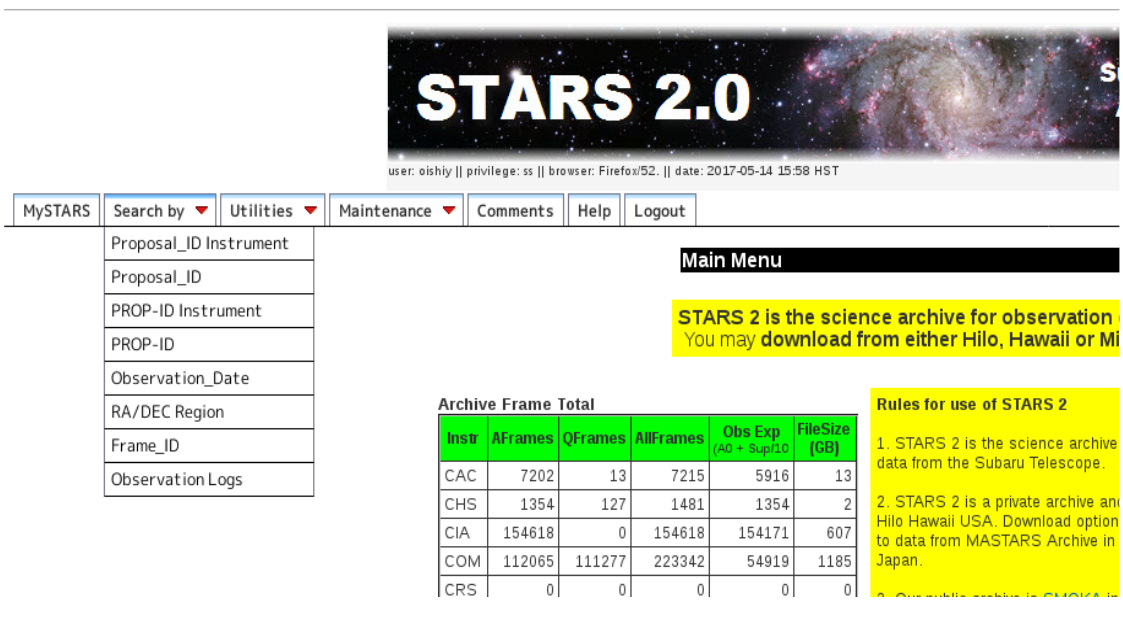
図1:STARS Search by¶
プロポーザル ID を選択すると、図2 のようなページになります。
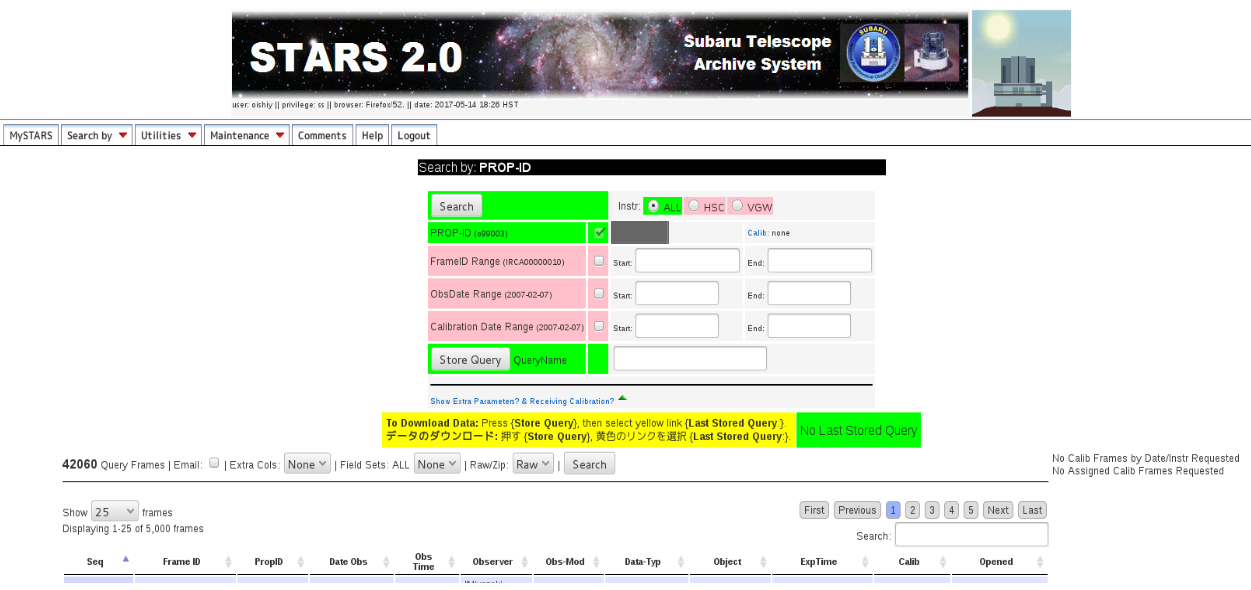
図2:STARS Search¶
選択したプロポーザル ID 全てのデータが欲しい場合は、Store Query を押してデータのリクエストを行います。特定のフレームのデータのみが欲しい場合は、FrameID Range で必要なデータの範囲を指定して適当な QueryName をつけ、Store Query を押してください。
Store Query を押すと「STARS2 Stored Query」というメールが届きますので、メールのインストラクションに従ってダウンロードして下さい。 もしくは、MySTARS タブからでもダウンロード用スクリプト (S2Query.tar) を取得できます。 MySTARS タブをクリックし、先程保存した QueryName を選択します。すると、One Query という名前のページが表示されます。 このページの一番下 (図3) から S2Query.tar を落として下さい。
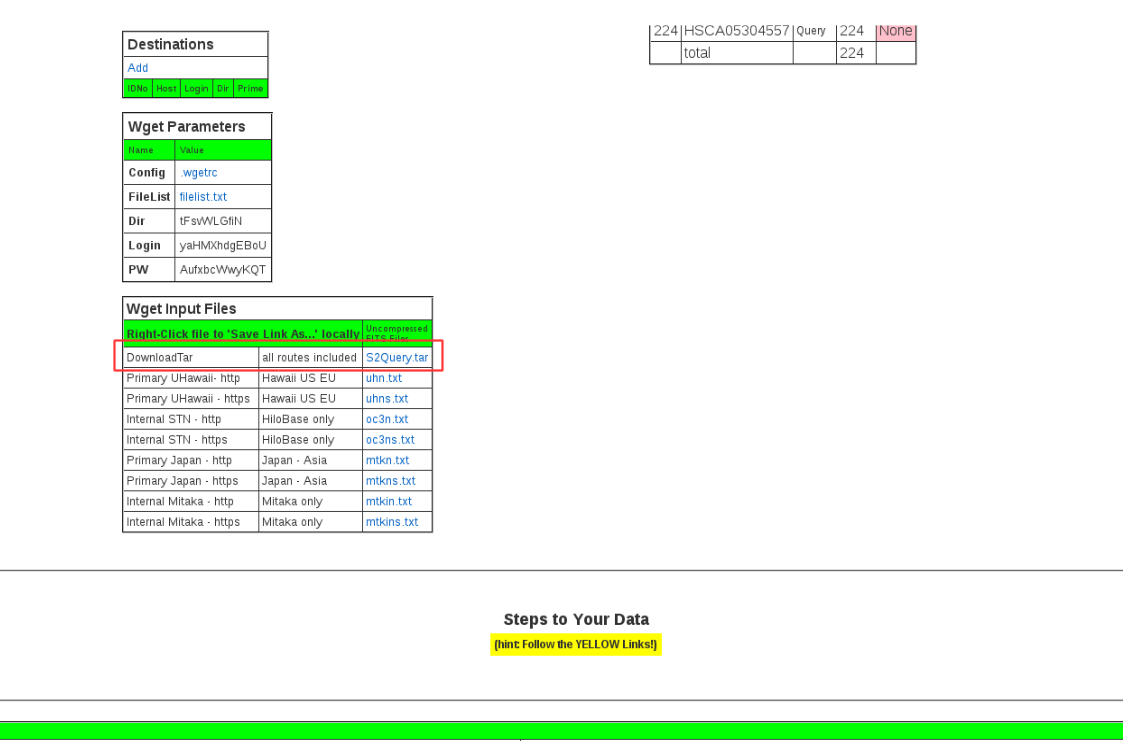
図3:STARS S2Query download¶
S2Query.tar ファイルを使って生データをダウンロードする方法を以下に示します。
# 生データを格納するディレクトリを準備 (rawdata というディレクトリを作りたい場合)
mkdir ~/rawdata
# S2Query.tar (~/Downloads にある場合) を rawdata ディレクトリに移動
cd rawdata
mv ~/Downloads/S2Query.tar .
# 展開
tar xvf S2Query.tar
# 生データをダウンロードするためのスクリプトが /zadmin 以下にあるので実行する
./zadmin/unpack.py
# スクリプトを実行するとネットワークの選択をする部分がありますので、御自身の環境にあったネットワークをお選びください。
ダウンロードが成功すると ~/rawdata の中に HSCA*.fits というファイルが見つかります。生データの名前については データ名について を参照下さい。
解析ディレクトリと各種ファイルの設置¶
生データのダウンロードが終了したら、次は解析ディレクトリや解析に必要なファイルの準備をします。 ここで行う事は、1) 解析ディレクトリの作成と _mapper ファイル作成、2) Brighter-Fatter カーネルの設定、3) アストロメトリ用カタログファイルへのリンク作成 です。 それぞれの詳細も含め、以下を確認して下さい。
解析ディレクトリの作成と _mapper ファイル作成¶
_mapper ファイルはデータがどの観測装置で取られたものかを示すものになります。
Warning
_mapper ファイルがないと解析が進まないので必ず作成してください。
# 必要な場合は、環境変数のリセット(マシンによっては環境変数をリセットしないとhscpipeのコマンドがうまく走らない場合があります)。hanacoでは不要。
unset LD_LIBRARY_PATH
# HSC pipeline セットアップコマンドを登録する。以上までは .bashrc等に書き込んでも良い。
source ~/opt/hscpipe/7.9.1/bashrc
# hscpipe setup コマンドで環境変数を設定
setup-hscpipe
# 以上までをログインの度に行う
# 解析用ディレクトリを作成 (ホームディレクトリの下に HSC というディレクトリを作り、そこを解析ディレクトリとする場合)
mkdir ~/HSC
# _mapper ファイルを作成
echo 'lsst.obs.hsc.HscMapper' > ~/HSC/_mapper
Brighter-Fatter カーネルの設定¶
hscPipe5 では、1次処理の際に行われる brighter-fatter 効果 (明るい星のような高カウントのピクセルから電荷が周囲に浸み出し、天体が広がって見える効果) を取り除くカーネルを自身の解析ディレクトリの下にコピーする必要があります。 バイナリパッケージをインストールし、create-rootdir.sh を実行した場合はこの作業も同時に行われています。 バイナリパッケージを使用しなかった場合、以下に方法を示します。
# BFKERNEL ディレクトリ (名前は必ず BFKERNEL にして下さい) を ~/HSC/CALIB の下に作成。
mkdir ~/HSC/CALIB/BFKERNEL
# brighter_fatter_kernel.pkl は $OBS_SUBARU_DIR/hsc/ の下にあるので、そこへ向けてリンクを張ります。
cd ~/HSC/CALIB/BFKERNEL
ln -s $OBS_SUBARU_DIR/hsc/brighter_fatter_kernel.pkl
アストロメトリ用カタログファイルへのリンク作成¶
hscPipe の CCD 解析では、1次処理の後にアストロメトリ較正、等級原点較正、天体の検出と測定を行います。 その際に外部のカタログファイルを参照します。
Note
hscPipe4 以前ではログインの度に setup astrometry_net_data コマンドを実行する必要がありましたが、hscPipe5 ではこのリンクを作成するだけで良いです。
# カタログファイル本体は ~/astrometry_data/ にあるとする。
# 解析ディレクトリの下に ref_cats ディレクトリを作成 (名前は必ず ref_cats にして下さい)。
mkdir ~/HSC/ref_cats
# ref_cats に移動し、リファレンスカタログへのリンクを張る。
cd ~/HSC/ref_cats
ln -s ~/astrometry_data/ps1_pv3_3pi_20170110
HSC用 transmission curveのセット¶
hscPipe 6以降では、自分でtransmission curveをセットしなければなりません。下記のコマンドを実行してください。
installTransmissionCurves.py ~/HSC
生データを解析レジストリに登録¶
STARS からダウンロードした生データを解析ディレクトリに配置し、レジストリに登録します。 使用するコマンドと使用例を紹介します。
# 解析ディレクトリの下に生データを配置し、レジストリを作成
# 生データは ~/rawdata の下にあり、current directory は ~/ であるとします。
# ディレクトリは絶対パスで指定。
ingestImages.py ~/HSC ./rawdata/*.fits --mode=link --create
# ingestImages.py [解析ディレクトリ] [登録したい fits ファイル名] --mode=[move,copy,link,skip] --create
# オプション
# --mode: データの配置方法。link の場合は生データへのリンクを作成します。
# --create: 新しくレジストリを作成する場合の引数。
データの配置、登録が完了すると、~/HSC の下に [Object Name], DARK, DOMEFLAT 等のディレクトリと registry.sqlite3 というレジストリができます。 [Object Name]ディレクトリや DOMEFLAT ディレクトリの構造は ~/HSC/[Object Name, BIAS, DARK, DOMEFLAT]/[dateObs]/[opinting]/[filter]/HSCA*.fits となっていると思います。
次に、SQL というデータベース言語を用いて、registry.sqlite3 の中身を確認してみましょう。
# レジストリを SQLite で読み込む
sqlite3 registry.sqlite3
# ヘッダ情報読み込み
sqlite> .header on
# データの表示
sqlite> SELECT visit, filter, field, count(visit)
...> FROM raw # レジストリから上記情報を選択
...> GROUP BY visit, filter, field; # visit > filter > field 順に並べて表示
# select filter, field, visit, count(visit) from raw group by visit; のように一行で書いてもよい。
# 結果が表示される
visit|filter|field|count(visit)
17636|HSC-Z|DOMEFLAT|112
17638|HSC-Z|DOMEFLAT|112
17640|HSC-Z|DOMEFLAT|112
17642|HSC-Z|DOMEFLAT|112
17644|HSC-Z|DOMEFLAT|112
17646|HSC-Z|DOMEFLAT|112
18336|HSC-I|DARK|112
18350|HSC-Y|DARK|112
.....
# 終了
sqlite> .q
SQL についてここでは詳しく説明しませんので、興味のある方は各自で調べるようにして下さい。
ここで見るべきポイントは count(visit) が 112 であることです。これは 112 枚分の CCD データが1つの visit に対して存在しているということです。 また、HSC-Z の DOMEFLAT visit は 17636 から 17646 というように、各 filter, field の visit がどれであるかが確認できます。
Warning
hscPipe では CCD データが足りないにもかかわらず、処理が進む場合があります。解析を始める前に、全てのデータセットで CCD 112 枚分あるかを確認をしましょう。
